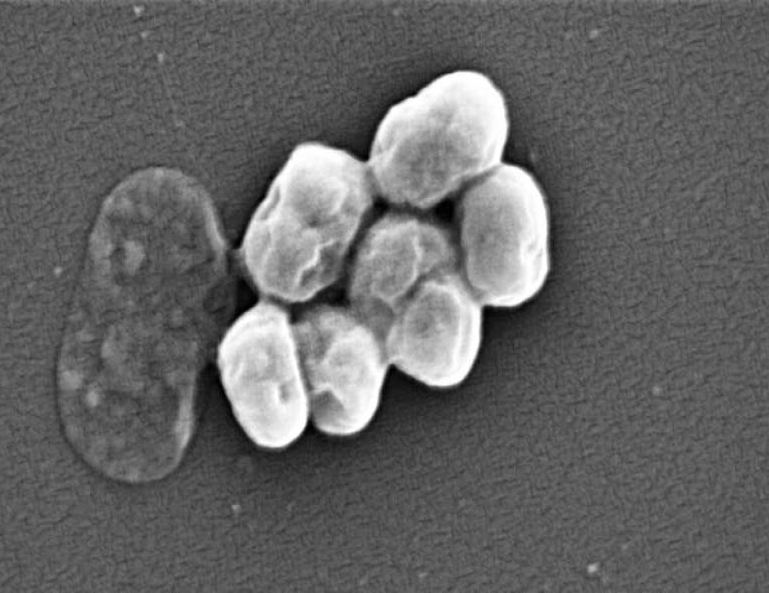
Con casi 5 millones de muertes relacionadas con la resistencia a los antibióticos cada año en todo el mundo, se necesitan con urgencia nuevas formas de combatir las cepas bacterianas resistentes. Investigadores de Stanford Medicine y la Universidad McMaster están abordando este problema con inteligencia artificial generativa. Un nuevo modelo, denominado SyntheMol (para sintetizar moléculas), creó estructuras y recetas químicas para seis nuevos medicamentos destinados a matar cepas resistentes de Acinetobacter baumannii, uno de los principales patógenos responsables de las muertes relacionadas con la resistencia a los antibacterianos. Los investigadores describieron su modelo y la validación experimental de estos nuevos compuestos en un estudio publicado el 22 de marzo en la revista Nature Machine Intelligence.
“Existe una enorme necesidad de salud pública de desarrollar rápidamente nuevos antibióticos”, afirmó James Zou, Ph.D., profesor asociado de ciencia de datos biomédicos y coautor principal del estudio. “Nuestra hipótesis era que existen muchas moléculas potenciales que podrían ser medicamentos eficaces, pero aún no las hemos fabricado ni probado. Por eso queríamos utilizar la IA para diseñar moléculas completamente nuevas que nunca se han visto en la naturaleza”.
Antes de la llegada de la IA generativa, el mismo tipo de tecnología de inteligencia artificial que subyace a grandes modelos de lenguaje como ChatGPT, los investigadores habían adoptado diferentes enfoques computacionales para el desarrollo de antibióticos. Utilizaron algoritmos para desplazarse por las bibliotecas de medicamentos existentes, identificando aquellos compuestos con mayor probabilidad de actuar contra un patógeno determinado. Esta técnica, que examinó 100 millones de compuestos conocidos, arrojó resultados, pero apenas arañó la superficie al encontrar todos los compuestos químicos que podrían tener propiedades antibacterianas.
“El espacio químico es gigantesco”, dijo Kyle Swanson, estudiante de doctorado en ciencias computacionales de Stanford y coautor principal del estudio. “La gente ha estimado que hay cerca de 1.060 posibles moléculas similares a fármacos. Por lo tanto, 100 millones no están ni cerca de cubrir todo ese espacio”.
Alucinando para el desarrollo de drogas
La tendencia de la IA generativa a “alucinar”, o inventar respuestas de la nada, podría ser una gran ayuda cuando se trata de descubrir fármacos, pero intentos anteriores de generar nuevos medicamentos con este tipo de IA dieron como resultado compuestos que serían imposibles de producir en el mundo real, dijo Swanson. Los investigadores necesitaban poner barreras alrededor de la actividad de SyntheMol, es decir, garantizar que cualquier molécula ideada por el modelo pudiera sintetizarse en un laboratorio.
“Hemos abordado este problema intentando cerrar la brecha entre el trabajo computacional y la validación en laboratorio húmedo”, dijo Swanson.
El modelo fue entrenado para construir fármacos potenciales utilizando una biblioteca de más de 130.000 componentes moleculares y un conjunto de reacciones químicas validadas. Generó no solo el compuesto final sino también los pasos que se siguieron con esos componentes básicos, brindando a los investigadores un conjunto de recetas para producir los medicamentos.
Los investigadores también entrenaron el modelo con datos existentes sobre la actividad antibacteriana de diferentes sustancias químicas contra A. baumannii. Con estas directrices y su conjunto de componentes básicos, SyntheMol generó alrededor de 25.000 posibles antibióticos y las recetas para elaborarlos en menos de nueve horas. Para evitar que las bacterias desarrollen rápidamente resistencia a los nuevos compuestos, los investigadores filtraron los compuestos generados solo para aquellos que eran diferentes de los compuestos existentes.
“Ahora no sólo tenemos moléculas completamente nuevas, sino también instrucciones explícitas sobre cómo producirlas”, dijo Zou.
Un nuevo espacio químico
Los investigadores eligieron los 70 compuestos con mayor potencial para matar la bacteria y trabajaron con la empresa química ucraniana Enamine para sintetizarlos. La empresa pudo generar de manera eficiente 58 de estos compuestos, seis de los cuales mataron una cepa resistente de A. baumannii cuando los investigadores los probaron en el laboratorio. Estos nuevos compuestos también mostraron actividad antibacteriana contra otros tipos de bacterias infecciosas propensas a la resistencia a los antibióticos, incluidas E. coli, Klebsiella pneumoniae y MRSA.
Los científicos pudieron probar más a fondo la toxicidad de dos de los seis compuestos en ratones, ya que los otros cuatro no se disolvían en agua. Los dos que probaron parecían seguros. El siguiente paso es probar los medicamentos en ratones infectados con A. baumannii para ver si funcionan en un cuerpo vivo, dijo Zou.
Los seis compuestos son muy diferentes entre sí y de los antibióticos existentes. Los investigadores no saben cómo funcionan sus propiedades antibacterianas a nivel molecular, pero explorar esos detalles podría arrojar principios generales relevantes para el desarrollo de otros antibióticos.
“Esta IA realmente está diseñando y enseñándonos sobre esta parte completamente nueva del espacio químico que los humanos simplemente no han explorado antes”, dijo Zou.
Zou y Swanson también están perfeccionando SyntheMol y ampliando su alcance. Están colaborando con otros grupos de investigación para utilizar el modelo para el descubrimiento de fármacos para enfermedades cardíacas y para crear nuevas moléculas fluorescentes para investigaciones de laboratorio.
Fuente: Tech Xplore.